Un modelo AlphaFold de Tmem81, una proteína de membrana implicada en la fusión del óvulo y el espermatozoide.Crédito: Google DeepMind/EMBL-EBI (CC-BY-4.0)
Durante casi una década, Andrea Pauli, bioquímico del Instituto de Investigación de Patología Molecular de Viena, ha estado intentando descubrir cómo se unen el espermatozoide y el óvulo.
En 2018, su laboratorio encontró una proteína en la superficie del pez cebra (danio rerio) huevos, llamados Bouncer, que eran esenciales para la fertilización. Pero el equipo de Pauli y otros tuvieron dificultades para demostrar cómo Bouncer reconocía los espermatozoides. Entonces ocurrió una revolución.
AlphaFold revela cómo se conectan el espermatozoide y el óvulo con todo detalle
Hace cinco años, a finales de noviembre de 2020, investigadores de Google DeepMind, con sede en Londres, presentaron AlphaFold2. La herramienta de inteligencia artificial para predecir estructuras de proteínas generó modelos 3D asombrosamente precisos que, en algunos casos, eran indistinguibles de los mapas experimentales. Dominar un desafío de predicción de estructuras a largo plazo.. La primera versión de AlphaFold se anunció en 2018, pero sus predicciones no fueron tan buenas como las de su sucesor, lo que limitó su impacto.
El 2021 liberación del código de AlphaFold2 y un base de datos que ha aumentado a cientos de millones de estructuras predichas significa que los científicos ahora pueden obtener una predicción confiable para casi cualquier proteína.
«Tener modelos para cualquier cosa ha tenido un impacto enorme», afirma Janet Thornton, bioinformática del Instituto Europeo de Bioinformática en Hinxton, Reino Unido, parte del Laboratorio Europeo de Biología Molecular (EMBL-EBI). «Es como la segunda venida de la biología estructural».
Descubrimiento rápido
Para el equipo de Pauli, el software arrojó luz sobre un camino que de otro modo nunca habrían encontrado. El modelo predijo que una proteína, llamada Tmem81, estabiliza un complejo de otras dos proteínas del esperma, creando un bolsillo al que Bouncer se puede unir.1. experimentos hizo una copia de seguridad de las predicciones de la herramienta. AlphaFold «acelera el descubrimiento», dice Pauli. «Lo utilizamos para cada proyecto».
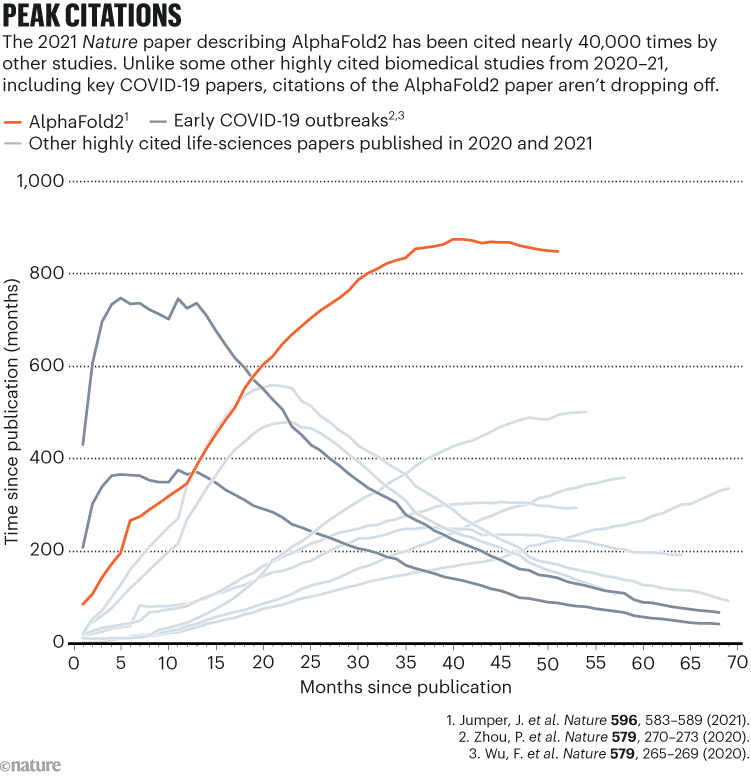
El artículo de su equipo sobre esto, publicado en 2024, es uno de los casi 40.000 artículos de revistas que citan el año 2021. Naturaleza artículo que describe AlphaFold22. A diferencia de muchos otros artículos biomédicos y de ciencias biológicas muy citados del mismo período, incluidos informes fundamentales sobre la pandemia de COVID-19, el interés en AlphaFold no parece estar desacelerando (consulte 'Pico de citas').
John Jumper de DeepMind, que compartió la mitad del Premio Nobel de Química 2024 con el director ejecutivo Demis Hassabis para desarrollar AlphaFold, dice que está “profundamente orgulloso” de lo útil que ha sido la herramienta para científicos como Pauli. «¿Cuándo alguien ganará uno de estos importantes premios porque utilizó AlphaFold?» se pregunta.
Parte del rápido impacto de AlphaFold2 se debe a su accesibilidad, dicen los investigadores. Google DeepMind puso el código subyacente y otros parámetros a disposición de los científicos de forma gratuita, y rápidamente les fue posible ejecutar el software a escala: esto es lo que hizo el equipo de Pauli.

Unos 3,3 millones de usuarios en más de 190 países han accedido a la base de datos AlphaFold (AFDB), alojada en EMBL-EBI y que contiene más de 240 millones de predicciones estructurales, que abarcan la mayoría de las proteínas conocidas. Más de un millón de usuarios del BAfD provienen de países de ingresos bajos y medios, incluidos China e India (ver 'Llamamiento global').
Revolución de la estructura de las proteínas
El campo en el que AlphaFold parece haber tenido su mayor impacto es la biología estructural. Los investigadores que utilizaron AlphaFold enviaron alrededor de un 50% más de estructuras de proteínas a un depósito de modelos experimentales, llamado Banco de datos de proteínas (PDB), que una 'línea de base' de investigadores de biología estructural que no utilizan AlphaFold, encuentra un estudio financiado por Google DeepMind estudio de los impactos de AlphaFold lanzado esta semana. El uso de AlphaFold2 también se asoció con tasas más altas de envíos de PDB que las de los investigadores que utilizan otros métodos «de frontera» en IA, biología estructural y predicción de la estructura de proteínas (ver «Acumulación de proteínas»).

Fuente: IA en la ciencia: evidencia emergente del impacto de AlphaFold2
Jumper dice que está especialmente satisfecho de que AlphaFold2, que fue entrenado utilizando datos de PDB — ha demostrado ser muy útil para deducir estructuras de proteínas. Las estructuras predichas pueden ayudar a los investigadores a dar sentido a los datos brutos generados por cristalografía de rayos X y microscopía crioelectrónica. «Me encanta que ayuda a las personas que nos proporcionaron los datos», añade Jumper.